Г.Г. ГОНЧАРЕНКО, С.А. ЗЯТЬКОВ
Представлены данные по кариотипу и геному домашней кошки F. catus. Рассмотрены микросателлитные локусы в качестве наиболее удобных генетических маркеров для дактилоскопии особей и пород F. catus. Охарактеризованы идентификационная панель, основанная на использовании 10 локусов с динуклеотидными повторами с вероятностью совпадения генотипа равной 10-10, а также 11-локусная панель с тетрануклеотидными повторами с вероятностью совпадения 10-6–10-8
.
Ключевые слова: ДНК, кошачьи, цепная реакция, особь.
The karyotype and genome of the domestic cat F. catus are described. A forensic genotyping panel of 10
dinucleotide STR loci (power of discrimination 10-10) and 11 tetranucleotide STR loci (power of
discrimination 10-6-10-8) for F. catus is characterized.
Keywords: DNA, cat, chain reaction, individual.
Кариотипы и геномы. Раннее в цитогенетических исследованиях было установлено,
что кариотип домашней кошки Felis catus L. состоит из 38 хорошо различимых хромосом –
18 пар аутосом и пары половых хромосом XY. Хромосомы домашней кошки ясно отличаются размером и положением центромеры. У них различают три крупных субметацентрических
(A1-A3), четыре субтелоцентрических (B1-B4) и две метацентричеких (C1-C2) хромосомы.
Имеются также мелкие хромосомы – четыре субметацентрические (D1-D4), три метацентрические (E1-E3), две акроцентрические (F1-F2) и субметацентрическая X и маленькая Y хромосома [1]–[4]. Изображение 18 аутосом и половых хромосом Х и Y домашней кошки F. catus виде идиограммы представлено на рисунке 1.
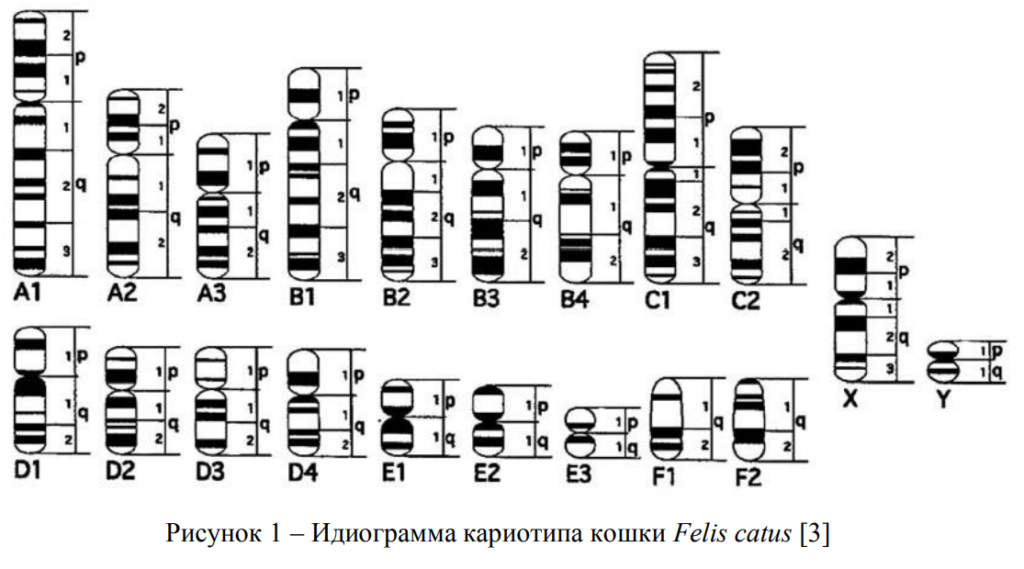
Успешное развертывание исследований по проекту «Геном кошки» (2002-2007) [5], [6]
и ранее завершенному проекту «Геном человека» (2000-2003) [7], [8] дало возможность анализировать на молекулярном уровне как отдельные структурные гены, так и любые другие
участки ДНК в геномах этих видов. Установлено, что гаплоидный геном человека (23 хромосомы) содержит 3,2 × 109 нуклеотидных пар ДНК и около 32 тыс. генов, а геном кошки (19
хромосом) – 2,7 × 109 н. п. и более 20 тыс. генов.
В последние годы на основе данных по структуре генома стремительно развиваются методы
ДНК-идентификации, позволяющие проводить дактилоскопию отдельных особей, генетическую
паспортизацию пород и установление видовой принадлежности особей семейства Кошачьи.
Характеристика микросателитных локусов. Одним из наиболее удобных и широко
используемых маркеров для ДНК-идентификации являются микросателлиты. Это особый
класс ДНК-маркеров, представляющих собой фрагменты ДНК с большим количеством – до
ста и выше тандемно повторяющихся идентичных «мотивов». Мотивом является короткая
последовательность из нескольких (от двух до восьми) пар нуклеотидов обычно называемая
«повтором» [9].
В зависимости от длины повтора микросателлиты классифицируют на локусы с ди-, три-,
тетра-, пента-, и гексануклеотидными повторами. Таким образом, микросателлитом, или микросателлитным локусом (STR-локусом, Short Tandem Reapets), называют – участок ДНК, расположенный в конкретной хромосоме и содержащий короткие тандемные повторы.
Аллели микросателлитного локуса отличаются друг от друга числом повторов и, как
следствие, длиной. Микросателлитные локусы высокополиморфны – т. е. для каждого из них
имеется много аллелей. Например, локус FCA149, локализованный в хросмосоме В1, домашней кошки содержит динуклеотидные повторы ТГ. В популяциях F. catus обнаружено 6
аллелей этого локуса (с числом повторов от 13 до 18). Нуклеотидная последовательность
фрагмента ДНК, содержащего повторы ТГ, и прилежащие к повторам справа и слева (фланкирующие) последовательности ДНК представлены на рисунке 2. Из рисунка хорошо видно,
что данный аллель локуса FCA149 имеет 17 повторов ТГ. Поэтому формула данного локуса
записывается как FCA149 (ТГ)17.

Рисунок 2 – Фрагмент ДНК, содержащий ТГ повторы локуса FCA149 и фланкирующие участки
Микросателлитные фрагменты выявляют методом полимеразной цепной реакции (ПЦР),
обеспечивающим амплификацию – многократное увеличение копий данного фрагмента ДНК.
Синтез этого фрагмента инициируется ДНК-затравками в виде пары праймеров, синтетических олигонуклеотидов, комплементарных нуклеотидным последовательностям на границах
исследуемого фрагмента. Так как микросателлитные аллели короткие и вместе с праймерами
обычно не превышают 200–300 п. н., то даже сильно поврежденный биологический материал
может содержать полные копии исследуемого фрагмента ДНК, обеспечивая их успешную амплификацию. Именно по этой причине ПЦР микросателлитов оказался особенно важным для
судебно-медицинских исследований. Для исследуемого микросателлитного локуса конструируют такую пару праймеров, чтобы комплементарные им фланкирующие участки ДНК были
высокоспецифичны, т. е. отсутствовали в других участках генома. Длина праймеров должна
быть не менее 20–30 п. н., их 3′-концы не должны быть комплементарными друг другу [10].
Интересным примером микросателитного локуса является протоонкоген вируса саркомы кошки fes/fps, который расположен в длинном плече хромосомы 15 человека и содержит
тетрануклеотидные повторы ATTT в интроне 5 этого гена. Повторы ATTT и определяют
микросателлитный локус, обозначаемый как FES/FPS. В популяциях человека обнаружен ряд
аллелей локуса FES/FPS с числом повторов от 7 до 15. На рисунке 3 дана нуклеотидная последовательность фрагмента интрона 5 гена fes/fps, между позициями 4631 и 4800, где располагаются повторы ATTT (полная длина гена fes/fps превышает 12 тыс. пар нуклеотидов).
Формула STR-локуса FES/FPS записывается как (ATTT)11, поскольку в наиболее характерном аллеле содержится 11 повторов ATTT (на рисунке 3 выделены жирным). На этом же рисунке подчеркнуты участки ДНК для пары праймеров, которые успешно используются при
амплификации аллелей локуса FES/FPS.
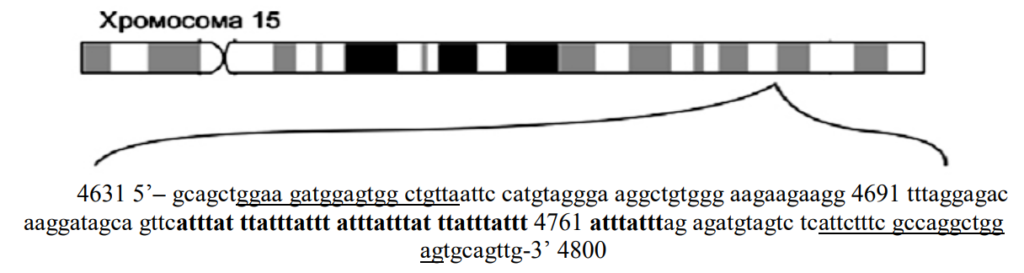
Рисунок 3 – Фрагмент ДНК, содержащий АТТТ повторы микросателлитного локуса FES/FPS и фланкирующие участки, расположенные в интроне 5 протоонкогена вируса саркомы кошки fes/fps, локализованного в длинном плече хромосомы 15 человека
Необходимо подчеркнуть, что в последние десятилетия были разработаны эффективные методы анализа микросателлитов с использованием праймеров, меченных флуоресцентными красителями, с последующей детекцией продуктов реакции с помощью автоматических секвенаторов ДНК [11].
Панель микросателитных локусов для идентификации особей F. Catus. За последние годы было установлено, что микросателлитные локусы у всех исследованных видов являются высокополиморфными, включая и микросателлиты F. catus, с десятками аллелей в каждом локусе и высокими темпами мутирования [12]–[14].
Поскольку микросателлитные локусы являются селективно-нейтральными, они не подвержены действию естественного отбора. Комбинация аллелей таких локусов является уникальной характеристикой каждой особи.
Интересно отметить, что уже в первой работе, посвященной разработке методов дактилоскопии кошек на основе микросателлитов Менотти-Раймонд с соавторами [15] использовали 10 микросателлитных локусов с динуклеотидными повторами. В предыдущем разделе нашей статьи приведен пример (рисунок 2) для одного локуса FCA 149. В нем обнаружено
6 аллелей, содержащих динуклеотидный мотив ТГ с количеством повторов от 13 до 18. Следовательно, при 6 аллелях в популяциях F. catus по этому локусу будет 21 различный генотип. Если использовать для идентификации кошек кроме локуса FCA 149 еще один локус
также с 6 аллелями, это позволит различать (21 × 21) 441 генотип. В работе МеноттиРаймонд и др. [15] использовалось 10 микросателлитных локусов, количество аллелей в каждом из которых варьировало от 5 до 10. Это означает, что количество возможных многолокусных генотипов по этим 10 генам у кошек составляет более 10 миллиардов (1010). Иными словами только у одной особи F. catus из 10 млрд. генотип при использовании этого набора локусов, может совпасть с какой любо другой особью. Таким образом, разработанная по 10 микросателлитным локусам идентификационная панель позволяет проводить точную генетическую дактилоскопию любой особи F. catus.
В дальнейшем исследователи предложили для дактилоскопии кошек использовать набор из 11 микросателлитных локусов, содержащих тетрануклеотидные повторы [16]. Тетрануклеотидный мотив, хромосомная локализация, количество аллелей, а также оптимальный набор праймеров для амплификации этих 11 локусов приведены в таблице 1. Данная идентификационная панель позволяет проводить точную генетическую дактилоскопию для 28 пород F. catus с вероятностью совпадение генотипов у двух особей равной 10-6, а для непородистых – 10-8 [16]. Эта панель прошла, этапы сертификации и получила статус стандарта для ДНК-тестирования в криминалистических лабораториях США.
Необходимо добавить, что для контроля происхождения и индивидуальной идентификации кошек Международным обществом генетики животных (ISAG) с 2006 года рекомендована также идентификационная панель, использующая систему маркеров, основанную на анализе 9 микросателлитных локусов с ди- и тетрануклеатидными повторами [17], [4].
Таблица 1 – Характеристика 11 тетрануклеотидных микросателлитных локусов иллюстрирующие
ДНК идентификационную панель
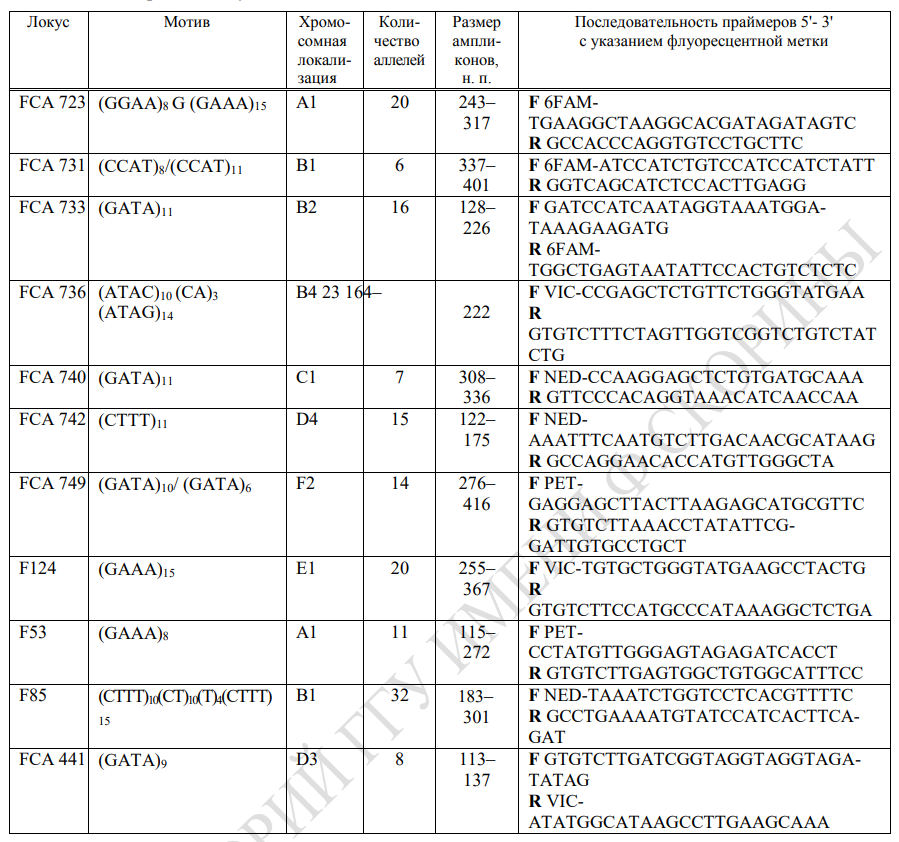
Таким образом, в данной статье представлены данные по кариотипу и геному домашней кошки F. catus. Рассмотрены микросателлитные локусы в качестве наиболее удобных
генетических маркеров для дактилоскопии особей и пород F. catus. Охарактеризованы идентификационная панель, основанная на использовании 10 локусов с динуклеотидными повторами с вероятностью совпадения генотипа равной 10-10, а также 11-локусная панель с тетрануклеотидными повторами с вероятностью совпадения 10-6
–10-8
.
Работа проводилась в рамках тем ГПНИ 16-14 и ГПНИ 16-32, выполняемых в рамках
государственных программ «Биотехнологии» и «Природопользование и экология».
Литература
- Jones, T.C. San Juan Conference on karyotypes of Felidae. Special Report / T.C. Jones // Mammal.
Chromos. Newslett. – 1965. – Vol. 15. – P. 121–122. - О’Брайен, С. Генетика кошки / С. О’Брайен, Р. Робинсон, А.С. Графодатский, А.В. Таранин. –
Новосибирск: Наука, 1993. – 212 с. - Cho, K.W. A proposed nomenclature of the domestic cat karyotype / K.W. Cho, H.Y. Youn,
H. Satoh, T. Watari, H. Tsujimoto, A. Hasegawa // Cytogenet Cell Genet. – 1997. – V. 79. – P. 71–78. - Lyons, L.A. Genetic testing in domestic cats / L.A. Lyons // Molecular and Cellular Probes. – 2012. –
doi:10.1016 / j.mcp.2012.04.004. - O’Brien, S.J. The Feline Genome Project / S.J. O’Brien, M. Menotti-Raymond, W.J. Murphy,
N. Yuhki // Annu. Rev. Genet. – 2002. – V. 36. – P. 657–686. - Pontius, J.U. Initial sequence and comparative analysis of the cat genome / J.U. Pontius, J.C.Mullikin,
D.R. Smith, K. Lindblad-Toh, S. Gnerre, M. Clamp, J. Chang, R. Stephens, B. Neelam, N.
Volfovsky, A.A. Schaffer, R. Agarwala, K. Narfstrцm, W.J. Murphy, U. Giger, A.L. Roca, A. Antunes, M.
Menotti- Raymond, N. Yuhki, J. Pecon-Slattery, W.E. Johnson, G. Bourque, G. Tesler, S.J. O’Brien //
Genome Res. – 2007. – V. 17. – P. 1675–1689. - Initial sequencing and analysis of the human genome / International Human Genome Sequencing
Consortium // Nature. – 2001. – Vol. 409. – P. 860–921. - Venter, J.C. The sequence of the human genome / J.C. Venter [et al.] // Science. – 2001. –Vol. 291.
– P. 1304–1351. - Tautz, D. Notes on the definition and nomenclature of tandemly repetitive DNA sequences/
D. Tautz // DNA Fingerprinting: State of the Science, Basel, Switzerland, 1993. – P. 21–28. - Животовский, Л.А. Микросателлитная изменчивость в популяциях человека и методы ее
изучения / Л.А. Животовский // Вестник ВОГиС. – 2006. – № 1, Т. 10. – С. 74–96. - Ziegle, J.S. Application of automated DNA sizing technology for genotyping microsatellite loci /
J.S. Ziegle, Y. Su, K.P. Corcoran // Genomics. – 1992. – V. 14. – P. 1026–1031. - Jeffreys, A.J. Spontaneous mutation rates to new length alleles at tandem repetitive hypervariable
loci in human DNA / A.J. Jeffreys, N.J. Royle, V. Wilson, Z. Wong // Nature. – 1988. – V. 332. – P. 278-281. - Kelley, R. Spontaneous mutation at the hypervariable mouse minisatellite locus Ms6-hm: flanking
DNA sequence and analysis of germline and early somatic mutation events / R. Kelley, M. Gibbs, A.
Collick, A.J. Jeffreys // Proc. R. Soc. Lond. B. – 1991. – V. 245. – P. 235–245. - Henderson, S.T. Instability of simple sequence DNA in Saccharomyces cerevisiae / S.T.
Henderson, T.D. Petes // Mol. Cell. Biol. – 1992. – V. 12. – P. 2749–2757. - Menotti-Raymond, M. Genetic Individualization of Domestic Cats Using Feline STR Loci for
Forensic Applications / M. Menotti-Raymond, V.A. David, J.C. Stephens, L.A. Lyons, S.J. O’Brien //
Journal Of Forensic Sciences. – 1997. – V. 42 (6). – P. 1039–1051. - Menotti-Raymond, M. An STR Forensic Typing System for Genetic Individualization of
Domestic Cat (Felis catus) Samples / M.A. Menotti-Raymond, V.A. David, L.A. Lyons, L. Wachter,
J.M. Butler, S.J. O’Brien // J.Forensic.Sci. – 2005. – Vol. 50 (5). – P. 1061–1070. - Lipinski M.J. An international parentage and identification panel for the domestic cat (Feliscatus)
/ Y. Amigues, M. Blasi, T.E. Broad, C. Cherbonnel, G.J. Cho, S. Corley, P. Daftari, D.R. Delattre,
S. Dileanis, J.M. Flynn, D. Grattapaglia, A. Guthrie, C. Harper, P.L. Karttunen, H. Kimura, G.M.
Lewis, M. Longeri, J.C. Meriaux, M. Morita, R.C. Morrin-O’donnell, T. Niini, N.C. Pedersen, G. Perrotta,
M. Polli, Rittler, R. Schubbert, M.G. Strillacci, H. Van Haeringen, W. Van Haeringen, L.A. Lyons // Anim
Genet. – 2007. – V. 38 (4). – P. 371–377.
Гомельский государственный
университет им. Ф. Скорины Поступила в редакцию04.03.2016




